精密化の開始
X-Buildの起動
“QUANTA”→“Applications”→“X-Build”を選択(図2.1.1)。
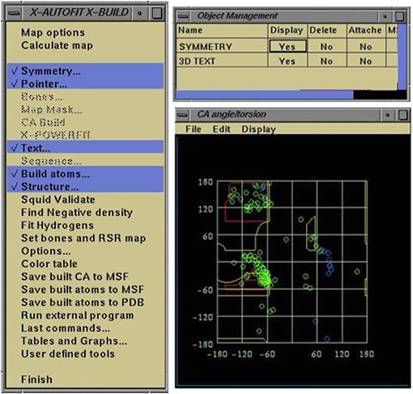
⇒X-Build起動後、“X-AUTOFIT X-BUILD”、“Object Management”、“CA angle torsion”の各パレットが新たに表示(図2.1.2)。
“QUANTA”画面に電子密度が表示。

(図2.1.1) “QUANTA”メニュー“Applications” |
(図2.1.2) “X-Build”の起動・表示パレット
|
“X-AUTOFIT X-BUILD”パレットに表示された、各メニューをクリックすると、新たにパレットが表示される(図2.1.3)。
各パレットの機能を用いて構造の修正(グラフィックイメージの精密化)を進める。
“Symmetry” 〜結晶学的情報の入力〜
補足:ver.upに伴い(?)結晶系に関するパラメータ入力は自動化されているかもしれません。
以下、不要な情報かもしれませんが、念のため記述を残しておきます。(2009年10月 記)
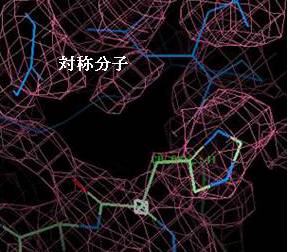
“Symmetry”パレット(図2.1.3a)で結晶系を設定することで、対称分子を表示させることが可能となる。
対称分子の表示は、電子密度がどの分子に由来するかを判断する上で重要である。
手順は以下の通り。
-
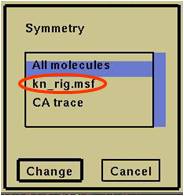
“Symmetry”パレット“Define symmetry”をクリック。
⇒“Symmetry”が表示(図2.1.4)。
-
開いているmsfファイル(“kn_rig.msf”)を選択し、“Change”。
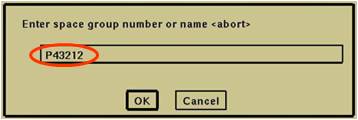
⇒“Enter space group number or name ”が表示(図2.1.5)。
-
空間群を入力し、“OK”。
⇒“Enter dimensions for ・・・”が表示(図2.1.6)。
-
格子定数を入力し、“OK”。
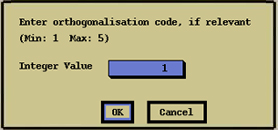
⇒“Enter orthogonalisation code, if relevant”が表示(図2.1.7)。
-
“Integer Value”は1のまま、“OK”。
⇒近傍の対称分子が青で表示される(図2.1.8)。
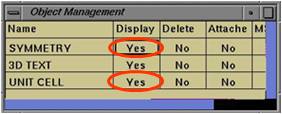
*対称分子は“Object Management”(図2.1.9)、“SYMMETRY”・“Display”の“Yes”をクリックすると未表示になる(“No”に変わる)。
(図2.1.4) “Symmetry”
|
(図2.1.5) “Enter space group number or name”
|

(図2.1.6) “Enter dimensions for …”
|
(図2.1.7) “Enter orthogonalisation code, …」
|
(図2.1.8) 対称分子の表示 |
(図2.1.9) “Object Management”
|
以上の操作を行うと、次の“参考:空間群・対称分子の表示”に示した機能を使用できる。
参考:空間群・対象分子の表示
通常の精密化の過程で、これらの機能を用いることはそれほど無いと思うが、参考までに記述。
“Unit cell”
クリックすると、画面に空間群が表示される(図2.1.10a)。
*“Object Management”パレット“UNIT CELL”の“Display”を“No”で未表示になる。
“CA Filled Cell”および“Filled Cell”
クリックすると、空間群内の分子配置が表示される。
・“CA Filled Cell”:対称分子の主鎖構造のみが表示。
・“Packing Diagram”:全原子が表示(図2.1.10b)。
*“Symmetry”パレット“Delete Sym. Ob.”をクリックすると表示か消える。
“CA Packing Diagram”および“Packing Diagram”
クリックすると、近接する対称分子が表示される。
・“CA Packing Diagram”:対称分子の主鎖のみが表示(図2.1.10c)。
・“Packing Diagram”:対称分子の全原子が表示。
*“Symmetry”パレット“Delete Sym. Ob.”をクリックすると表示が消える。

(図2.1.10) 空間群・対称分子の表示
|
Pointer”〜目標とするアミノ酸残基への移動〜
基本的に“QUANTA”画面の中心には、ポインターが表示されており、
“Pointer”パレット (図2.1.3b)でそれを動かすことが可能。
ポインターを目標とするアミノ酸残基へ移動させると、その残基が画面中心に表示されることになる。
平たく言えば、目的のアミノ酸残基へ画面を動かすために用いるパレット。
それぞれの機能は以下の通り。
“Go to pointer”
クリックすると、ポインターのある位置が画面の中心になる。
“Place at next residue”および“Place at previous residue”
それぞれクリックすると、前後のアミノ酸残基へポンターが移動する。
“Place by atom”
目的のアミノ酸残基へポインターが移動する。
-
“Place by atom”をクリック。
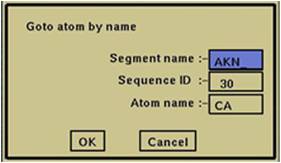
⇒“Goto atom by name”が表示(図2.1.11)。
-
“Segment name”:セグメント名(参考:“Segment name”・原子の情報)を入力。
“Sequence ID”:目的のアミノ酸残基の番号を入力。
“Atom name”:目的の原子名を入力(通常、CA)。
-
“OK”
⇒目的のアミノ酸残基(例では30残基目)のCα原子にポインターが移動。
(図2.1.11) “Goto atom by name”
|
ポインターの手動
Shift+Controlキーを押しながらマウスを動かすと、任意の位置へポインターを移動可能。
その後、“Go to pointer”により、ポインターの移動先が画面中心になる。
参考:“Segment name”・原子の情報
“Segment name”は構造(pdb)ファイル上で、タンパク質、リガンド、水などの個々の分子に与えられる名前
(例えばXY2量体、水分子が含まれるpdbファイルでは、X鎖に“A”、Y鎖に“B”、水分子に“W”とセグメント名が与えられていることが多い)。
ただし、各分子に与えられている“Segment name”は、pdbファイルごとに若干異なっている(例では、タンパク質のセグメント名が“AKN”)ことがあるので、
各分子のセグメント名はpdbファイル上で確認しておく。
また“QUANTA”画面上で適当な原子をクリックすると、その原子の名前、セグメント名、残基番号が確認できる(図2.1.12)。
加えて、クリックした原子の情報は“QUANTATextport”(図2.1.13)(参考“QUANTA Testport”)にも表示される。
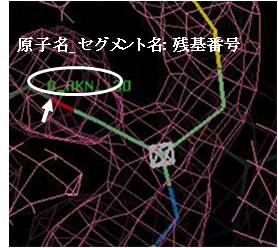
(図2.1.12) 原子の情報 |
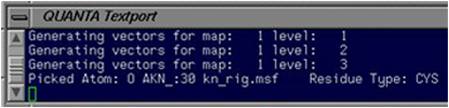
(図2.1.13) “QUANTA Textport”
|
参考:“QUANTA Textport”
“QUANTA Textport”(図3・5)は、QUANTA起動中に表示されているターミナルlであり、各パレットで操作した詳細が全て記載されている。
原子の情報の他、実行したコマンドも“QUANTA Textport”上で確認できる。
Back