X-Ligandは、リガンド分子に該当する電子密度を探索しモデルを挿入、さらにその構造の修正ができる。
起動は以下の通り。
-
“QUANTA”メニュー“Applications”→“X-Ligand”を選択(図4.2.1)。
⇒ “X-Ligand”パレットが表示され、“QUANTA画面”に対称分子が青表示(図4.2.2)。
 (図4.2.1) “QUANTA”メニュー“Applications” |

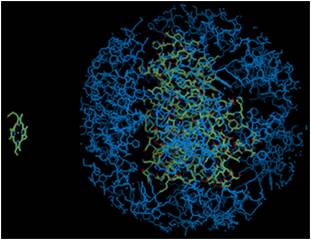 (図4.2.2) X-Ligandの実行 |
リガンドの探索
条件を設定後、リガンドが当てはまる電子密度の探索を行う。
-
“X-Ligand”パレット“Change search setting”をクリック。
⇒ “Ligand search parameter”が表示(図4.2.3)
-
パラメータ入力後、“OK”。
-
“X-Ligand”パレット“Search for ligands”をクリック。
⇒ 探索された電子密度にリガンドの分子モデルが挿入され、“X-Ligand”パレット上の機能が使用可能(実線表示)となる(図4.2.4)。
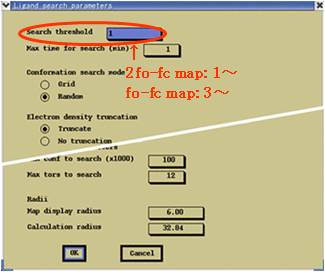 (図4.2.3) “Ligand search parameter” 図ではパレットの一部を省略。 “Search threshold”欄に電子密度にあったシグマレベルを入力。 |

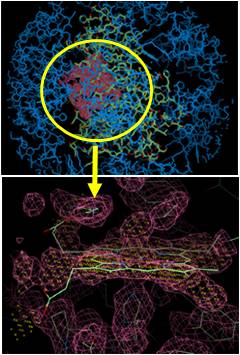 (図4.2.4) “Search for ligands”の実行 |
挿入された位置(電子密度)に問題がなければ、次の修正を行う。
リガンドの修正
“Search for ligands”実行により、新たに実線表示されたプログラムを用いて、リガンド分子モデルの修正を行う。
-
“X-ligand”パレット“Search conformations”をクリック。
⇒電子密度へのフィッティングが行われ、候補構造が白表示される(図4.2.5)。
加えて、“Search comformations”以下の各機能が実線表示され、使用可能になる(図4.2.6)。
-
“X-ligand”パレット上の修正プログラム(図4.2.6)を用いて、リガンドの分子モデルの修正を行う。
*各修正プログラムの機能は、X-Buildの“Build Atoms”の 各プログラムに類似しているので、参考のこと。
ちなみに図4.2.7は“Refine all”を実行した例。
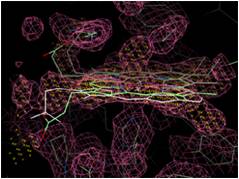 (図4.2.5) “Search comformations”の実行 |
 (図4.2.6) 修正プログラム |
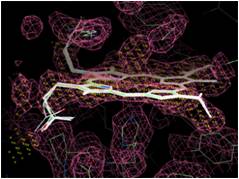 (図4.2.7) “Refine all”の実行 |
なお、リガンド分子の修正はX-Buildでも実施可能。
結果の保存・X-Ligandの終了
結果を保存し、X-Ligandを終了する。
-
“X-ligand”パレット“Save ligand to MSF”をクリック。
⇒ ファイル名の入力画面が表示される(図4.2.8)。
-
適切なファイル名(例では“Ligand-Ref.msf”)を入力し“OK”。
⇒修正後のリガンドの分子モデルが、新規msfファイルとして保存。
既存の蛋白質・リガンドに加え、新規保存したモデルが“Molecule Manegement”に表示される(図4.2.9)。
-
“X-ligand”パレット“Exit”をクリック。
⇒X-Ligandが終了する。
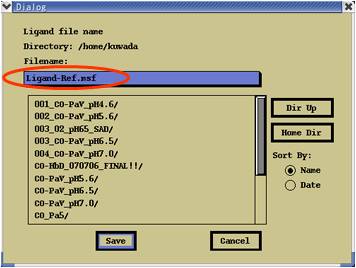 (図4.2.8) リガンド分子の選択 |
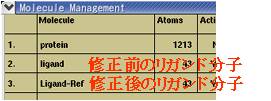 (図4.2.9) “Molecule Manegement” |
以上でX-Ligandによる、リガンド分子の挿入および修正が終了。
なお、図4.2.9に示す様に、修正前のリガンド分子の構造ファイルも読み込まれている。
不必要であれば、修正前のリガンド分子の構造ファイルのみを“Close”する(図4.2.10)。
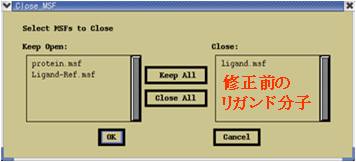 (図4.2.10) “Close MSF” 修正前のリガンド構造ファイルを選択(クリック)し“OK”。 |