リガンドの挿入を行うためには、元となるリガンドの構造ファイルが必要となる。
リガンドの構造ファイル(pdbファイル)は、各種分子モデルの情報サイト“HIC-Up”から入手するか、
既に目的のリガンドを含む構造が“Protein Data Bank”に登録されているならば、それをダウンロードしリガンドのみのファイルに編集すればよい。
“HIC-Up”からの入手
-
“HIC-Up”Topページ左のサブパネルに表示されている“General”・“Search HIC-Up”をクリック。
⇒ “HIC-Up - Google Search !”が表示。
-
“Text to search for: ”欄にキーワード(リガンド名)を入力後、“Google Search !”をクリック。
⇒ 検索結果が表示。
-
検索結果から適切なものを選択し、クリック。
⇒ リガンドに関する詳細が表示される。
-
“Coordinates”から、“PDB file”もしくは“Clean PDB file”をクリックし、ダウンロードする。
“Protein Data Bank”からの入手〜編集
-
目的のリガンドを含むpdbファイルを通常の手順で、ダウンロード。
-
リガンド以外の情報を削除する(text ファイルとして編集可)。
*下の例の様にリガンドの“ATOM”行のみに編集。“TER”“END”行は残す。
ATOM 1213 FE LIG A1001 7.178 4.824 -1.247 1.00 10.99 A FE
ATOM 1214 CHA LIG A1001 4.704 2.531 -0.555 1.00 17.17 A C
…(中略)…
ATOM 1254 O1D LIG A1001 0.490 2.024 0.433 1.00 32.26 A O
ATOM 1255 O2D LIG A1001 -1.245 3.250 -0.079 1.00 27.17 A O
TER 1256 LIG A1001
END
X-Ligandを実行するには、以下、3つのファイルが必要である。
-
精密化中の蛋白質分子の構造ファイル(pdb or msf 形式)
-
精密化で用いている電子密度ファイル(map or mbk 形式)
-
入手したリガンド分子の構造ファイル(pdb形式)
それぞれ、以下の手順で読み込む。
蛋白質分子の構造ファイル
ファイル形式に合わせた通常の手順、つまりpdb形式なら“Import single structure”、
msf形式なら“Open”で良い。
電子密度ファイル
ファイル形式に合わせた通常の手順、つまりmap形式なら“Import”、
mbk形式なら“Add a map”で良い。
なお、電子密度ファイルは、リガンドの電子密度が正しく表示されるものであれば良い(2fo-fc map, fo-fc map、など)。
リガンド分子の構造ファイル
リガンド分子のpdbファイルも、蛋白質のpdbファイル同様、“Import single structure”によりmsf形式に変換するが、追加作業がある。
-
“QUANTA”メニュー“File”から“Import single structure” を選択。
⇒ “Select File to Import”が表示。
-
リガンド分子のpdbファイルを選択し、“Import”をクリック。
*ここまでは通常の蛋白質分子のpdbファイルの“Import”手順と同じ。以降の手順が追加される。
⇒ リガンド分子の選択画面が表示される(図4.1.1)。
-
適切なもの(類似したタイプの高分子)を選択し“OK”。
⇒ 結晶系入力に関する確認画面が表示(図4.1.2)
-
“NO”をクリック。
⇒ 画面表示する分子モデルの確認画面が表示(図4.1.3)
-
“Add this to your previous selection”を選択し“OK”。
⇒“QUANTA”画面に蛋白質に加え、リガンドの分子モデルが表示される(図4.1.4)。
“Molecule Management”には蛋白質及びリガンドのファイルが表示。
|
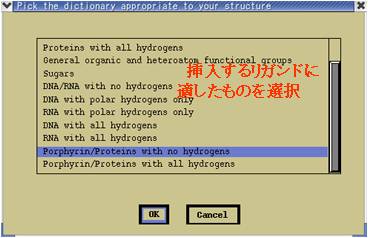
(図4.1.1) リガンド分子の選択
リストから適切な高分子の種類(核酸、糖、など)を選択。
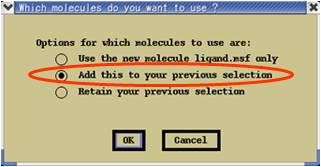
(図4.1.3) 分子表示の確認
既に読み込まれている蛋白質モデルに追加して表示させる。
|
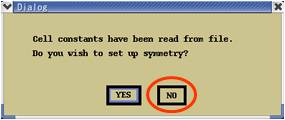
(図4.1.2) 結晶系入力の確認
通常、蛋白質分子の結晶系を用いるので再入力の必要はない。
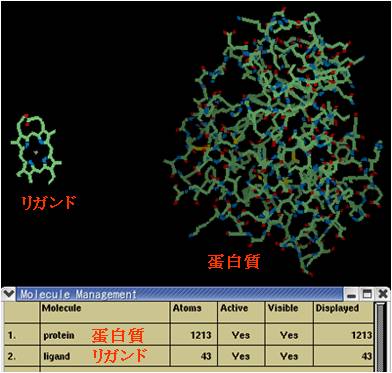
(図4.1.4) リガンド分子の表示
|
以上でX-Ligand実行の準備が整う。
なお、構造ファイルを読み込んだ時点では、リガンド分子が本来位置すべき場所とは、全く異なる場所に表示される(図4.1.4)。
この位置のずれを、X-Ligandで修正し、正しい配置の分子モデルを作成する。



